-- 논문보다는 개인적인 생각을 바탕으로 서술하였습니다--
어떤 학습을 목표로 하는지에 따라서 다르겠지만 마이크로바이옴 데이터를 학습시키기 위해서 무조건 포함되어야 하는 것은 균주의 정량 또는 유무에 대한 정보입니다. 16s rRNA gene 을 증폭시켜 해당 부분을 시퀀싱한 데이터를 얻었다고 가정할 때 해당 데이터를 처리하는 방법은 생각보다 다양할 수 있습니다.
그리고 머신러닝 학습의 특성상 최소 백명 또는 천명 단위의 데이터를 필요로 하는데 동일한 조건으로 해당 데이터를 생산하는 것은 쉽지 않습니다.
이전에 마이크로바이옴 데이터를 사용하여 조산을 예측하고자 머신러닝 경진대회와 비슷한 것이 열렸었습니다.
Microbiome Preterm Birth DREAM Challenge: Crowdsourcing Machine Learning Approaches to Advance Preterm Birth Research
Globally, every year about 11% of infants are born preterm, defined as a birth prior to 37 weeks of gestation, with significant and lingering health consequences. Multiple studies have related the vaginal microbiome to preterm birth. We present a crowdsour
www.ncbi.nlm.nih.gov
해당 대회에는 3909 명의 샘플을 가지고 머신러닝을 수행했는데 모델을 학습시키기에 충분한 데이터 개수라고 생각합니다. 다만, 해당 데이터들이 모두 동일한 방법으로 생산된 것은 아닙니다. 그럼에도 그런 것들은 충분히 무시하고 수행해도 문제 없다고 여겨지는 부분인 것 같습니다.
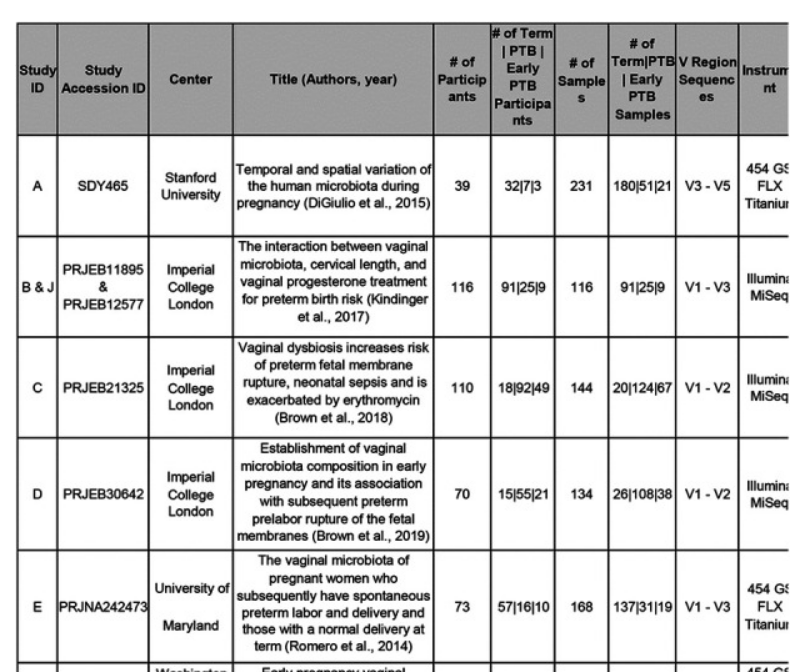
보시면 증폭한 variable region 의 위치도 다를 뿐더러, 시퀀싱 수행한 기기도 다양한 것을 볼 수 있습니다.
아무래도 저렇게 많은 수의 데이터를 확보하려면 지금으로써는 저것들을 통일한다는 것이 현실적으로 어렵기 때문이 아닌가 싶습니다.
그럼에도 이후에 조금 더 생각해봐야하는 부분이 있습니다.
가장 단순하게 생각하여, 아래와 같은 데이터를 학습시켰다고 가정해보겠습니다.
(물론 피험자에 대한 성별, 나이, 인종 이외에도 추가적인 정보가 있겠지만 필수적으로 사용되는 시퀀싱 데이터만 나타내겠습니다.)
| Sample | feature1 | feature2 | feature3 ..... | group |
| sample1 | ? | ? | ? | obesity |
| sample2 | ? | ? | ? | healthy |
| sample3 | ? | ? | ? | obesitiy |
물론 feature 를 정량한 데이터를 넣어줘야 할 것 입니다.
다만, 어떤 형태의 데이터을 넣어야 샘플 끼리 또는 run 끼리 비교 가능한가 대해 고민해봐야 합니다.
feature table

dada2 수행 이후 feature table -> frequency 를 확인할 수 있습니다. frequency 가 의미하는 것은 각 feature (300 bp 의 merged reads) 가 몇 번 확인되었는지 입니다.
하나의 run 내에서는 frequency 를 이용해서 비교할 수 있겠지만 (시퀀싱 과정에서 무작위성이 추가되어 어느 정도의 편향이 있을 수는 있습니다) 다른 run 끼리 비교할 때 sequencing depth 의 차이 등으로 정확한 비교는 다소 어려울 수 있습니다. 물론 rarefying 을 수행하여 sequencing depth 를 맞춰줄 수 있겠지만, 데이터의 손실이 기계학습 모델의 성능에 얼마나 영향을 미칠까에 대해서도 확인해봐야 합니다.
또한, 해당 데이터를 사용한다면 어느 분류까지 collapse 할지에 대한 부분도 확인해볼 필요가 있습니다. collapse 하지 않고 feature 를 그대로 학습시킬 수도 있겠지만 기계학습의 목표에 따라서 species, genus, family 등으로 각각 collapse 할 필요도 있습니다. 물론 최초 feature 를 그대로 사용했을 때 기계학습 모델에 많은 feature 를 넣어줄 수 있으므로 모델의 복잡성을 높여줄 수도 있지만 0 을 많이 포함하고 있는 특성 상 0 을 줄여주기 위한 collapse 가 좋은 방법일 수 있습니다.
위와 어느 정도 연결되는 부분인데 frequency 데이터는 생각보다 편차가 큽니다. 물론 RF 모델은 숫자 데이터의 스케일링 없이도 좋은 성능을 보이지만 SVM, decision tree 등은 생각보다 스케일링의 효과가 괜찮다고 생각합니다. 하여 그런 부분도 충분히 고려해봐야 합니다.
해당 feature table 을 가지고 분석하다보면 결국에는 relative abundance 형태의 파일을 얻게 됩니다. 아래와 같은 compositional data 라고 할 수 있는데요, 이런 형식의 데이터는 모델을 학습시키는데 적합할까요?
| feature1 | feature2 | feature3 | feature4 | Sum | |
| Sample1 | 0.25 | 0.25 | 0.25 | 0.25 | 1 |
| Sample2 | 0 | 0 | 0 | 1 | 1 |
| Sample3 | 0.333 | 0.333 | 0 | 0.333 | 1 |
마이크로바이옴 데이터의 이런 특징은 독립적인 데이터가 아니기 때문 기본적인 통계 기법들 (PCA 등) 을 사용하지 못하는 원인 중 하나입니다. 따라서 머신러닝을 수행할때 relative abundance 를 사용하고자 한다면 logratio transformation 등의 변환을 수행한 뒤 학습시키는게 일반적인 것 같습니다. 아래의 논의를 참고하였습니다.
Compositional Data and Machine Learning
One of my features is compositional of nature, represented by a vector [p1, p2, p3]. Each vector represents an emotion and each vector sums up to unity. Eg: [anger=0.10, sad=0.10, happy=0.80]. 1) ...
stats.stackexchange.com
오늘은 다소 개인적인 생각을 가지고 포스팅하여 사진보다는 글로써 많은 부분 작성하였습니다. 이제 도전해보는 단계라서 객관적인 내용을 포스팅하려면 조금 더 많은 공부가 필요할 것 같습니다.
해당 내용 관련해서 많은 의견이나 도움주시면 감사하겠습니다.
'[머신러닝]' 카테고리의 다른 글
| 마이크로바이옴 + 대사체 데이터 기계학습 (0) | 2024.01.21 |
|---|---|
| [머신러닝] q2-sample classifier (0) | 2023.12.17 |
| [머신러닝] CNN 을 사용한 마이크로바이옴 데이터의 분류 (T2D vs healthy) (1) | 2023.11.26 |
| 실제 데이터로 머신러닝 모델 만들기 : 2 (1) | 2023.09.17 |
| 실제 데이터로 머신러닝 모델 만들기 : 1 (0) | 2023.09.16 |