최근 후성유전학과 관련해 많은 연구들이 진행되고 있습니다. 그중에서 가장 활발하게 연구되는 부분은 DNA Methylation 인 듯합니다.
DNA Methylation 은 cytosine의 5번 탄소에 메틸기가 결합하여 DNA 염기서열이 유지되면서 유전자 발현 등의 일부 기능의 변화를 매개하는 대표적인 DNA modification 입니다. ( Adenine 의 Methylation 기전도 존재합니다.)
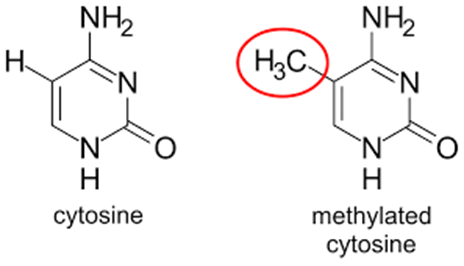
장내 마이크로바이옴 관련해서도 이러한 메틸화와의 상관관계를 연구하는 논문들이 출판되고 있는데 그 중심에는 DNA Methlyation 을 촉매하고 유전자 발현을 조절하는 효소인 DNA methyltransferases (DNMTs) 가 있습니다.
해당 효소는 장내 존재하는 일부 영양성분의 가용 여부에 따라 그 활성이 결정되는데 그러한 일부 영양성분들이 장내 미생물 군집의 대사 활동에 의해 영향을 받기 때문입니다.
DNA 메틸화를 위해 메틸기를 제공하는 6-methyltetrahydrofolate 의 합성에 장내 미생물의 대사물질인 folate, vitamin B12, betaine, choline 등이 사용되기 때문에 결국에는 DNA 메틸화 발생은 장내 미생물 군집의 구성과도 간접적으로 연결되어 있다고 말할 수 있습니다.
특히, folate (엽산) 의 생산은 Lactobacillus, Bifidobacterium 등에 의해 매개되고 있어 이러한 특정한 미생물의 정성, 정량적인 존재가 DNA 메틸화의 발생에 중요한 영향을 끼치게 됩니다.
또한, 장내 미생물에 의해 생산되는 대표적인 대사물질인 butyrate 를 포함한 SCFA (단쇄지방산) 중 일부가 DNA 메틸화 과정에 영향을 미치는데 이는 DNMTs 의 하향 조절과 연관이 있으며 탈메틸화를 일으킨다는 연구결과 또한 발표되었습니다.
그 외에도, DNA 메틸화와 장내 미생물 군집과의 연관성은 다양한 연구를 통해 입증되고 있지만 한 가지 요인이 아닌 다양한 요인이 관여하며 영향을 주고받아 정확한 기작을 확인하기 위해서는 더 많은 연구가 필요한 상황입니다.
DNA 메틸화는 박테리아 또는 박테리오파지에서 자주 나타나는데 DNA 염기서열의 변화가 없기에 특수한 시퀀싱이나 질량분석을 통해서 확인하는 것이 일반적입니다.
시퀀싱 분야에서는 일반적으로 Illumina 의 플랫폼들이 사용되는데 DNA 메틸화 등 DNA modification 을 확인하기 위해서는 PacBio, Nanopore 의 플랫폼을 사용합니다.
1. PacBio Single-Molecule Real-Time (SMRT) Sequencing
PacBio 의 SMRT Sequencing 은 대표적인 long-read sequencing 기법으로 long read DNA 를 원형으로 만들고, 해당 주형 DNA 를 zero-mode waveguides (ZMWs) 라고 불리는 수백 개의 웰이 포함된 칩에서 시퀀싱 됩니다..
주형 DNA 가 ZMWs 의 바닥에 고정된 뒤, 형광 표지된 염기서열이 DNA 중합효소에 의해 주형 DNA 에 합성되는데, 이때 합성된 염기 서열의 정보를 빛의 파장을 통해 구별합니다.
이 과정에서, 형광 표지된 염기서열이 변형된 염기서열에 결합될 때의 시간과 변형되지 않은 염기서열에 결합될때의 시간에 차이가 존재하는데 이 작은 시간의 차이를 구별함으로써 변형된 염기서열을 탐지할 수 있습니다.
이 시간의 차이를 “interpulse durations” (IPDs) 라고 정의하고 이러한 고유한 IPDs ratio 에 따라 해당 염기서열이 어떤 방식으로 변형되었는지 확인할 수 있습니다.
이 방식은 고유한 물질의 검출시간, 이온화된 성분의 분자량 등을 통해 어떠한 분자인지를 확인하는 질량분석기를 통한 물질의 확인과도 유사한 부분이 있습니다.

PacBio 에서 제공하는 SMRT Link software 를 사용하여 이러한 IPDs 정보 (.HDF5) 를 염기서열 정보 (.fasta) 로 변환할 수 있으며 Welch’s t test 를 기반으로 검정합니다
.
2. Nanopore sequencing
Nanopore sequencing 기법의 특징은 나노 스케일의 단백질 포어를 염기서열이 통과할 때 발생하는 전기적 신호를 구분하여 염기서열을 해독하는데, 메틸화된 염기와 메틸화되지 않은 염기의 전기적 신호의 차이는 10 pA 미만이지만 이러한 작은 차이를 해독하여 변형된 염기서열을 탐지할 수 있습니다.
다만, 인접한 염기서열이 서로의 신호에 영향을 미치는 ‘neighbouring effect’ 에 의해 탐지하는 것이 복잡해지고 특히, 새로운 변형을 해독하는데 어려움이 있지만 최근 몇몇 소프트웨어에서는 이러한 ‘이웃 효과’ 를 감안하여 염기서열의 변형을 탐지할 수 있도록 만들어져 있습니다.
3. 시퀀싱 이후 DNA 메틸화를 탐지하는 과정의 요약

위의 두 가지 시퀀싱을 통하면 4mC, 5mC, 5hmC, 6mA 와 같은 메틸화 기반의 염기서열 변형을 감지할 수 있지만 이는 아직까지는 제한적입니다.
기존에 알려져 있는 염기서열의 변형을 탐지하는 데에는 무리가 없지만 더욱 복잡하고 새로운 변형을 탐지하기는 어려우며 이러한 것들은 시퀀싱보다는 크로마토그래피와 질량분석기를 기반한 분석을 통해 알려진 것이 대부분입니다.
최근 분리한 미생물의 annotation 을 수행하다가 단백질 중에서 DNMT 가 있어서 관련 정보를 찾아보게 되었습니다.
한국에서도 PacBio long read sequencing 을 지원하는 회사가 몇몇 있는 것으로 알고 있는데 그 과정이 어렵지 않으면 한두 개 샘플 정도는 경험해 봐도 좋을 것 같다는 생각이 듭니다.
DNA 메틸화의 장내 미생물 군집과의 관련성이 입증되고 있어 추후 마이크로바이옴 분석에서도 결과를 해석할 수 있는 또 다른 요소가 될 수 있을 것 같습니다.
# reference
1) Sharma, Manvi, et al. "The epigenetic connection between the gut microbiome in obesity and diabetes." Frontiers in genetics 10 (2020): 453795.
2) Nielsen, Tue Kjærgaard, et al. "Detection of nucleotide modifications in bacteria and bacteriophages: Strengths and limitations of current technologies and software." Molecular Ecology 32.6 (2023): 1236-1247.
'bioinfo' 카테고리의 다른 글
| ECTyper - E. coli serotype identification (1) | 2024.04.27 |
|---|---|
| 인코렌탈 체험, 마이크로바이옴 데이터 간단 분석 (CLC Genomics Workbench Premium , incoRENTAL) (1) | 2024.04.22 |
| phylogeny for diversity analysis (qiime2 align-to-tree-mafft-fasttree) (0) | 2024.01.15 |
| Annotation (RAST) (0) | 2023.12.28 |
| Unifrac distance (microbiome) (0) | 2023.12.16 |