지난번 글에 이어서 LDA와 LEfSe에 대해 작성하겠습니다.
# statquest 유튜브를 참고하였습니다.
3. LDA (Linear discriminant analysis)
앞서 살펴봤던 PCA 와 PCoA와 유사한 방법인 것처럼 보이지만, LDA는 지도학습입니다. labeling 되어있지 않은 데이터를 가지고 수행하는 PCA와 PCoA와 달리, labeling 되어있는 데이터를 가지고 차원축소를 진행합니다. 그렇기 때문에, 각 그룹에 속하는 데이터의 seperatibility를 최대화하는 것에 목표가 있습니다.
PCA는 축에 정사영한 거리의 합이 최소가 되도록, 정사영하여 내린 점이 (0,0)으로부터 떨어진 거리가 최대가 되도록 축을 설정한다면, LDA는 그룹 내의 데이터 분산이 최소가 되도록, 그룹 간의 평균 차이가 최대가 되도록 설정한다. 이는 전체적인 데이터의 분산을 고려하는 PCA 와는 달리, 그룹끼리 더 잘 분리되도록 축을 설정하는 것이다.

아래 사진에서 PCA 와 LDA의 차이를 확인할 수 있다.


# 추가로, PCA 는 분포에 대한 가정이 없지만 LDA는 그룹 내 샘플들의 정규분포를 가정합니다. 이것이 LDA를 사용하기 까다롭게 하는 이유 중 하나입니다.
4. LEfSe (linear discriminant analysis effect size)
LEfSe 에 대한 논문에서의 설명은 다음과 같습니다.
해당 논문에서 제안하는 LEfSe는 고차원의 클래스 간의 비교를 지원하며 특히, metagenomic analyses에 중점을 둡니다. LEfSe는 통계적 유의성을 검증하는 표준검정에 생물 할 적 일관성 및 effect relevance를 고려한 검정을 결합하여 클래스 간 차이를 설명하는 데에 가장 유력한 특징 (organisms, clades, OTUs, genes, functions)을 판별합니다.
LEfSe는 간단하게 그룹 간의 어떤 taxa 가 유의한 차이를 보이는지 알려주는 도구입니다. 오늘은 LEfSe를 수행했을 때, 내부에서 어떤 식으로 동작하는지를 중점적으로 알아보겠습니다.

해당 논문의 fig6 을 확인하면 이를 알 수 있습니다.
step 1. taxa 의 relative abundance를 가지고 클래스 간에 kruskal-walis를 수행하여 유의한 것 (p-value < 0.05)을 선택합니다.
(KW, Wilcoxon 을 쓰는 이유는 metagenomnic data 가 relative abundance로 이루어 있기에 모수검정의 가정인 normal population을 위반하기에 비모수검정을 사용했다고 합니다.)
step 2. kruskal-walis 수행 이후에는 사후검정이 필요하므로 step 1 에서 유의했던 클래스의 모든 서브클래스끼리 Wilcoxon을 수행하여 어떤 서브클래스에서 유의한 차이가 있는지를 확인합니다.
step 3. step 2 번까지 수행하여 얻은 유의한 feature에 대해서, 클래스를 종속 변수로, feature values, subclass, subject values 를 독립변수로 하는 형식의 데이터를 작성합니다.
(이 부분에서 feature values, subclass, subject values 가 정확하게 무엇인지는 조금 더 알아봐야 할 것 같습니다.)
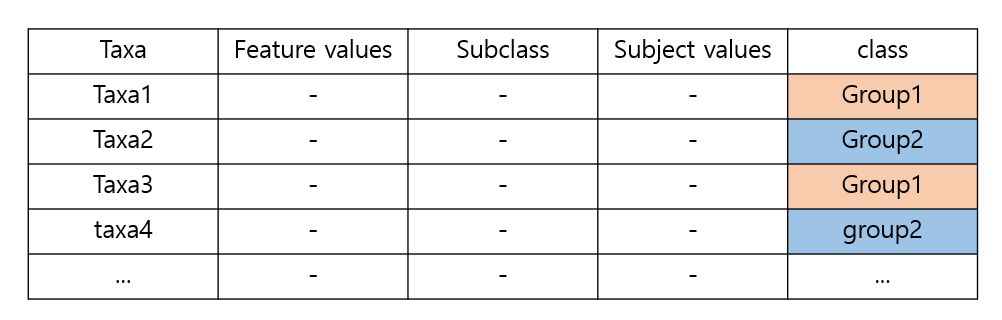
이것을 가지고 LDA 를 수행한 후 first linear discriminant axis를 사용, 데이터를 가장 분리도가 높도록 분리하고 LDA score 계산합니다. (LDA score는 사용자가 2,0, 3.0 등에서 선택할 수 있습니다.)

공부하고있는 입장이라 많이 부족하네요. 잘못된 부분에 대해 수정이나 의견 많이 부탁드리겠습니다.
# reference
1) https://www.youtube.com/watch?v=azXCzI57Yfc&t=754s
2) https://www.youtube.com/watch?v=9SuIE_tGF-g
3) Segata, Nicola, et al. "Metagenomic biomarker discovery and explanation." Genome biology 12 (2011): 1-18.
'bioinfo' 카테고리의 다른 글
| Sequencing quality (Illumina Miseq) : 1 (0) | 2023.09.01 |
|---|---|
| Sequencing quality : Phred quality score (illumina Miseq) (0) | 2023.08.28 |
| 차원축소 (PCA, PCoA, LDA) in microbiome data 와 LEfSe : 1 (microbiome) (0) | 2023.08.21 |
| differential abundance tools for microbiome data (상대적 풍부도) (0) | 2023.08.20 |
| OTU 와 ASV, 어떻게 ASV 는 선택되는가 (microbiome) (0) | 2023.08.19 |