마이크로바이옴 분석을 하다 보면 미생물 구성만으로는 많은 것을 말하기가 어렵습니다. 물론 특정 균주에 집중할 수도 있지만 16S 데이터의 경우 genus level 까지의 한계도 있고 전체적인 군집의 구성에 대해서는 다양성 분석 정도를 제외하면 할 수 있는 것이 많지는 않습니다.
그래서 이후에 미생물 군집의 대사경로로 넘어가는 경우가 많은 것 같습니다. 실제 질량분석기로 측정해서 어떤 대사물질들이 생성되는지도 확인할 수 있지만 PICRUST2 라는 소프트웨어를 사용하면 16S 데이터를 기반으로 대사경로를 예측, 정량해 줍니다. 물론 정확도의 차이는 있겠지만 질량분석이 가격이 조금 나가기에 하기 전에 한번 확인하는 용도로 괜찮습니다.
해당 소프트웨어에 대해서는 잘 설명되어있는 글들이 많아 참고하시면 될 것 같습니다.
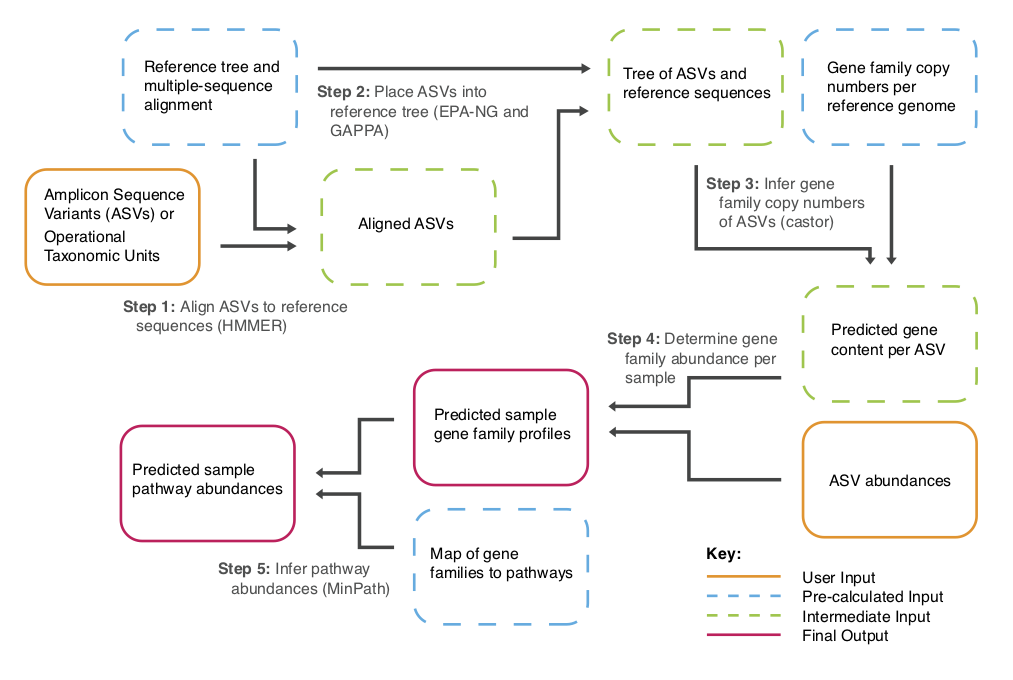
저도 PICRUST2 분석을 가끔 하는데 결과를 보면 가끔 결과에 나오는 대사경로들이 내가 확인한 미생물 군집의 구성과는 조금 다른 것처럼 보일 때가 있습니다.
저는 이전에도 포스팅했듯이 greengenes2 DB 를 이용해서 taxonomy assignment 를 하는데 만약 PICRUST2 에서 다른 DB 를 쓰면 내 taxa 와 PICRUST2 의 taxa 결과가 다를 거고 예측된 대사경로가 내가 확인한 미생물 군집의 구성과 일치하지 않을 것 같다는 생각이 들었습니다.
위의 Flowchart 를 보면 reference tree, MSA 에 대해 ASVs 를 alignment 하는 과정이 있기 때문에 충분히 그럴 수 있다고 생각했고 논문으로 가서 어떤 DB 를 사용했는지 확인해 보니 'IMG database (8 November 2017)' 을 사용한다고 합니다.
PICRUST2 github 를 들어가 봤는데 DB 에 대한 업데이트를 진행했다는 코멘트는 따로 없었습니다. 일단 위의 최초 DB 를 계속 사용하고 있는 것 같아서 DB 를 내가 원하는 것으로 변경할 수 있는지를 찾아봤습니다.
Frequently Asked Questions
Code, unit tests, and tutorials for running PICRUSt2 - picrust/picrust2
github.com
> github 의 FAQ 페이지에서 찾을 수 있었습니다. 질문 제목은
How can I run a custom or non-default database (such as the fungi 18S and ITS databases)?
입니다.
여기서는 16S 데이터베이스를 최신화하기 위한 것이 주목적은 아닌듯하고 18S, ITS 를 분석하기 위해 데이터베이스를 바꾸는 것을 예시로 들고 있습니다만 어쨌든 ASVs 를 alignment 하기 위한 DB 를 바꾸는 거니까 제가 이 부분을 보고 진행해도 괜찮을 것 같다고 생각했습니다.
가장 먼저 필요한 것은 greengenes2 (gg2) 데이터를 PICRUST2 와 호환될 수 있도록 변환해서 준비하는 것입니다. 아래의 4 개 파일이 필요하다고 합니다.
- A multiple-sequence alignment (with the extension .fna or .fasta and can optionally be gzipped)
- A tree in newick format (extension .tre)
- A hidden-markov model of the multiple-sequence alignment (extension .hmm)
- A modelfile output by RaXmL specifying the best parameters for the tree (extension .model)
익숙한 fast, fna, hmm 같은 형식도 있지만 .tre 나 .model 은 처음입니다. gg2 에서 제공하는 데이터들은 아래의 사진과 같습니다.

급하게 필요한 작업은 아니라서 시간을 두고 천천히 진행해보려고 합니다. 일단 필요한 파일을 바로 찾을 수 있었으면 좋았겠지만 없는 파일이 더 많아서 파일 형식들부터 하나씩 공부해 보면서 변환해야 할 것 같습니다.
이게 제가 생각하는 방식으로 작동하지 않을 수도 있습니다. 예를 들면, 데이터베이스를 gg2 로 바꿨는데도 PICRUST2 결과가 그대로 일수도 있는데 여러 가지 파일 형식에 대해서 공부해 볼 수 있는 계기가 될 것 같아서 시간 날 때마다 조금씩 공부해보려고 합니다. 이후에 이어서 작성하겠습니다.
# reference
1) Douglas, Gavin M., et al. "PICRUSt2 for prediction of metagenome functions." Nature biotechnology 38.6 (2020): 685-688.
'bioinfo' 카테고리의 다른 글
| AMRFinderPlus : 항생제 내성 유전자 탐지 (0) | 2024.08.13 |
|---|---|
| PICRUST2; Custom database 사용하기 - 2 (중단) (0) | 2024.08.03 |
| FDR correction in LEfSe (0) | 2024.05.12 |
| Beta diversity - Uniform Manifold Approximation and Projection (UMAP) (2) | 2024.05.07 |
| ECTyper - E. coli serotype identification (1) | 2024.04.27 |