PICRUST2 에서 커스텀 데이터베이스를 사용하기 위해 필요한 것은 아래와 같습니다.
- A multiple-sequence alignment (with the extension .fna or .fasta and can optionally be gzipped)
- A tree in newick format (extension .tre)
- A hidden-markov model of the multiple-sequence alignment (extension .hmm)
- A modelfile output by RaXmL specifying the best parameters for the tree (extension .model)
1. A multiple-sequence alignment (with the extension .fna or .fasta and can optionally be gzipped)
여기서 1 번 파일 (fna.gz) 는 아래 greengenes2 데이터베이스 에서 기본적으로 제공하므로 그대로 사용 가능합니다.
Index of /greengenes_release/current
ftp.microbio.me
2. A tree in newick format (extension .tre)
newick format 은 phylogenetic tree 를 컴퓨터에 인식시기 위해서 아래와 같은 규칙을 가지고 만들어진 파일입니다.
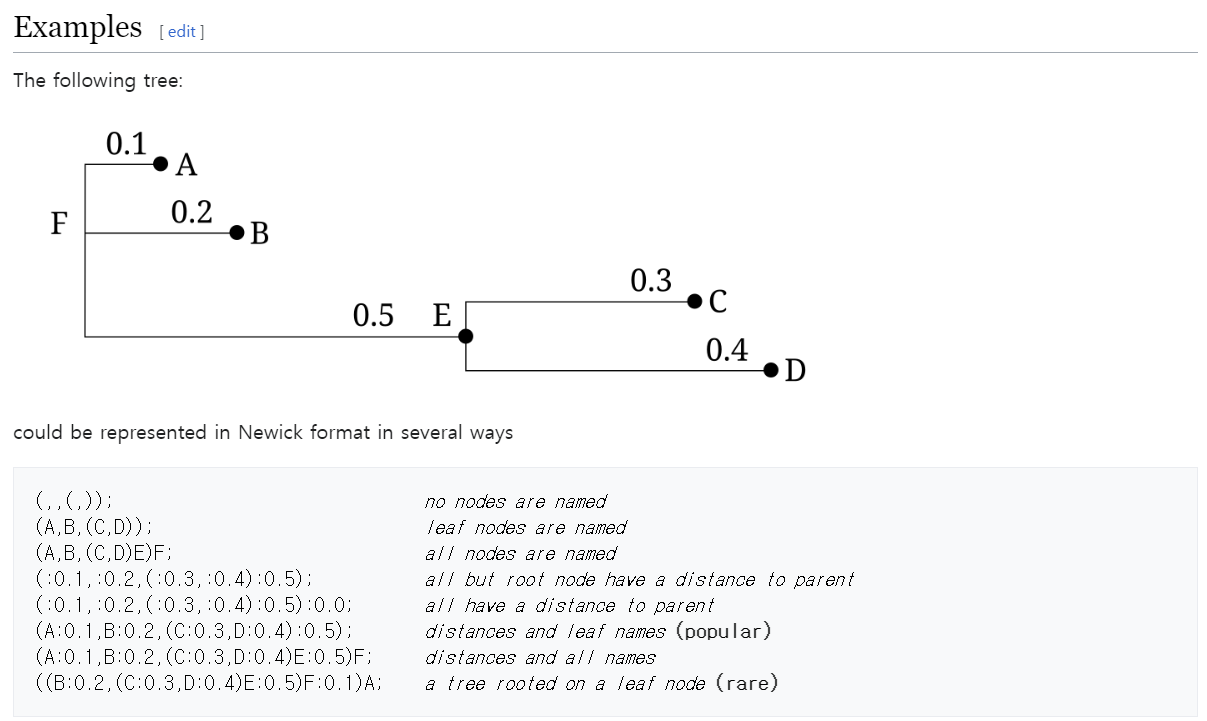
근데 이 newick 파일의 확장자가 .nwk 이고 greengenes2 데이터베이스에도 .nwk 확장자를 가진 파일은 많지만 .tre 파일 은 없어서 이 부분에 대해 확인해보니 .nwk 파일과 .tre 파일은 호환이 되고 확장자 명만 다른 것 같습니다.

그래서 일단 2022.10.phlyogeny.asv.nwk 파일의 확장자만 바꿔서 넣어볼 생각입니다.
3. A hidden-markov model of the multiple-sequence alignment (extension .hmm)
.hmm 확장자 파일 역시 제공되지 않습니다. 찾아보니 "hmmbuild' 를 사용해서 .hmm 파일을 만들어낼 수 있다고 합니다.
'hmmbuild' 는 hmmer 에서 사용할 수 있습니다. 아래와 같은 코드로 hmmbuiild 를 사용할 수 있습니다.
# 파일 다운로드
wget http://eddylab.org/software/hmmer/hmmer.tar.gz
# 압축해제 후 경로이동
tar -xvzf hmmer.tar.gz
cd hmmer-3.4
# 설치
./configure
make
sudo make install
# 설치 확인
hmmbuild -h
# 실행
hmmbuild seq.hmm seq.sto
+ MSA 파일은 Clustal Omega 를 사용해서 만들었으며 hmmbuild 가 input file 형식이 stockholm 일때만 인식하는 것 같아 .sto 형식으로 빼줬습니다.
+ 저는 V4 region 에 대한 분석을 하기 때문에 아래 파일에서 시퀀스를 빼서 사용했습니다.
* 2022.10.backbone.v4.fna.qza
In silico extracted V4 sequences from the backbone based on the EMP 16S primers
-----------------------------------------------------------------------------
위의 Clustal Omega 사용해서 multiple sequence alignment 형식으로 만드는 단계에서 계속 오류가 나네요,,ㅜ 확인해보니 greengene2 DB 의 용량이 너무 커서 처리를 못해주는 것 같습니다..
여기서 막혀버려서 일단 중단하고 나중에 다시 한번 도전해보겠습니다. 혹시나 PICRUST2 데이터베이스 바꾸는법 아시는분들이 계시다면 조언 부탁드립니다!!!
'bioinfo' 카테고리의 다른 글
| AMRFinderPlus : 항생제 내성 유전자 탐지 (0) | 2024.08.13 |
|---|---|
| PICRUST2; Custom database 사용하기 - 1 (중단) (0) | 2024.07.28 |
| FDR correction in LEfSe (0) | 2024.05.12 |
| Beta diversity - Uniform Manifold Approximation and Projection (UMAP) (2) | 2024.05.07 |
| ECTyper - E. coli serotype identification (1) | 2024.04.27 |