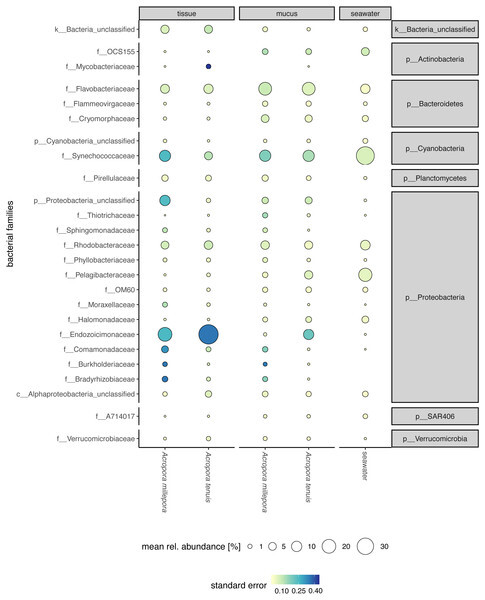
마이크로바이옴 관련 논문들을 많이 보면서 이 분야에 대해 잘 알지 못하는 사람들에게 보여줄 때는 어떻게 시각화하여 보여주는지가 중요하겠다는 생각을 많이 합니다. 마이크로바이옴 관련 논문이나 분석을 보면 아래의 세가지는 거의 항상 보입니다. alpha diversity, beta diversity, taxa plot (알파다양성을 나타내는 방식은 사실 모두 box plot 으로 상당히 익숙한 데이터 시각화라 제외하겠습니다.) 1. beta diversity 사실 베타다양성 같은 경우는 PCoA 사용하여 시각화하는데, PCoA 와 PCA 관련해서도 포스팅을 했었지만 거리를 기반으로 하느냐, 상관관계를 기반으로 하느냐의 차이만 존재하고 시각화가 되는 과정은 완전하게 동일합니다. 기본적으로 차원축소의 개념이기..